Librerías requeridas: dplyr, flextable, mixexp3 Comparación de métodos de imputación en diseños experimentales de mezclas
El presente documento corresponde a un apartado del trabajo titulado “Guía para la implementación de diseños experimentales en la industria farmacéutica”, presentado por Nathalia Cortés Duque como requisito para optar al título de Magíster en Ciencias - Estadística de la Universidad Nacional de Colombia.
4 Metodología
En los diseños de mezclas, se pueden presentar valores respuesta faltantes. Aunque existen diversos métodos de imputación, pocas veces se analiza de qué región del diseño experimental —ya sea de los vértices del simplex, de los puntos axiales o del centroide— proviene la ausencia de datos antes de seleccionar el procedimiento de imputación más apropiado.
La imputación constituye una herramienta estadística ampliamente utilizada para abordar este tipo de situaciones, especialmente cuando la falta de datos tiene un componente aleatorio. Siguiendo lo descrito por Wongoutong (2022) para diseños centrales compuestos, En este estudio se evaluó el efecto de un único valor de respuesta faltante en distintas zonas del diseño de mezclas (vértices, puntos axiales y centroide), aplicando diferentes métodos de imputación —media global, media parcial, regresión y K-vecinos más cercanos (KNN).
Imputación por la media global: consiste en reemplazar el valor faltante por la media de los datos disponibles.
Imputación por media parcial: el valor faltante se sustituye por la media de los valores no faltantes pertenecientes al mismo tipo de punto. Por ejemplo, si el dato perdido corresponde a un punto centrado en las caras, este se reemplaza por el promedio de los demás valores disponibles de tratamientos centrados en las caras.
Imputación por K vecinos más cercanos: el valor faltante se reemplaza por el promedio de los valores correspondientes a las observaciones más cercanas. Para ello, se definió una medida de proximidad entre los tratamientos; en este caso, se utilizó la distancia euclidiana. La clasificación de los puntos se encuentra en el Anexo 1.
Imputación por regresión: se ajusta un modelo de regresión con los datos completos y, a partir de este, se estima el valor faltante. En este trabajo se consideran los modelos de regresión propios de los diseños de mezclas, tanto en su forma lineal como cuadrática.
La caracterización de los tratamientos experimentales, conforme a la ubicación mostrada en la Figura 4.1, se presenta en la Tabla 12.2. El desempeño de los métodos de imputación se evaluó a partir de la generación de 1000 simulaciones de diseños de mezclas simplex-centroide, construidos bajo el modelo de regresión lineal característico de este tipo de diseños. En cada simulación se eliminó aleatoriamente un dato, lo que permitió comparar el valor imputado por cada método con el valor verdadero y, a partir de esta comparación, calcular el error. Se emplearon dos índicadores para cuantificar el error: el EAM (Error Absoluto Medio) y el RECM (Raíz del Error Cuadrático Medio), ambos calculados entre los valores reales y los imputados.
\[EAM = \frac{1}{n} \sum_{i=1}^{n} \left| y_i - \hat{y}_i \right|\]
\[RECM = \sqrt{\frac{1}{n} \sum_{i=1}^{n} \left( y_i - \hat{y}_i \right)^2}\]
donde \(y_i\) es el dato real, \(\hat{y}_i\) el dato imputado y \(n\) corresponde al número de observaciones disponibles utilizadas en el cálculo según el método de imputación.
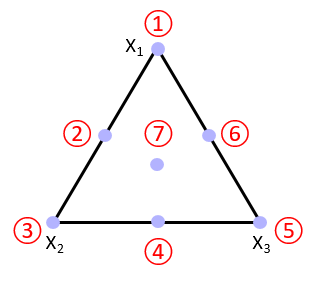
Tratamiento | x1 | x2 | x3 |
|---|---|---|---|
T1 | 1.00 | 0.00 | 0.00 |
T2 | 0.50 | 0.50 | 0.00 |
T3 | 0.00 | 1.00 | 0.00 |
T4 | 0.00 | 0.50 | 0.50 |
T5 | 0.00 | 0.00 | 1.00 |
T6 | 0.50 | 0.00 | 0.50 |
T7 | 0.33 | 0.33 | 0.33 |
T7 | 0.33 | 0.33 | 0.33 |
T7 | 0.33 | 0.33 | 0.33 |
5 Simulación de datos
Código
# Funcion para generar respuestas para diseños de mezclas
## des: dataframe que indica el diseño de mezclas
## sd: Desviación deseada para la generación del error
## mod: Tipo de modelo que se desea usar la para simulación de las respuestas, donde:
## 1: Modelo lineal
## 2: Modelo cuadrático
## 3: Modelo cúbico
gen_scheffe <- function(des,
sd = 1,
mod = c(1,2,3)) {
# explicar que es mod =1, 2 y 3
x1 <- des$x1; x2 <- des$x2; x3 <- des$x3 # las componentes
beta = round(runif(7, -10, 10),2)
# Modelo lineal
if (mod == 1)
{y_mean <- beta[1]*x1 + beta[2]*x2 + beta[3]*x3}
# Modelo cuadrático
if (mod == 2)
{y_mean <- beta[1]*x1 + beta[2]*x2 + beta[3]*x3 +
beta[4]*x1*x2 + beta[5]*x1*x3 + beta[6]*x2*x3}
# Modelo cúbico especial
if (mod == 3)
{y_mean <- beta[1]*x1 + beta[2]*x2 + beta[3]*x3 +
beta[4]*x1*x2 + beta[5]*x1*x3 + beta[6]*x2*x3 +
beta[7]*x1*x2*x3}
# Adición de error
yrta <- y_mean + rnorm(length(y_mean), mean = 0, sd = sd)
return(yrta)
}5.1 Modelo lineal
Se simulan 1000 modelos generados a partir de un modelo lineal canónico de Scheffé. A continuación, se presenta uno de ellos:
coefficients Std.err t.value Prob
x1 -8.685797 0.9906899 -8.767423 0.0001219853
x2 2.367515 0.9948362 2.379803 0.0547828282
x3 6.253771 0.9948362 6.286232 0.0007542658
Residual standard error: 1.237552 on 6 degrees of freedom
Corrected Multiple R-squared: 0.9423512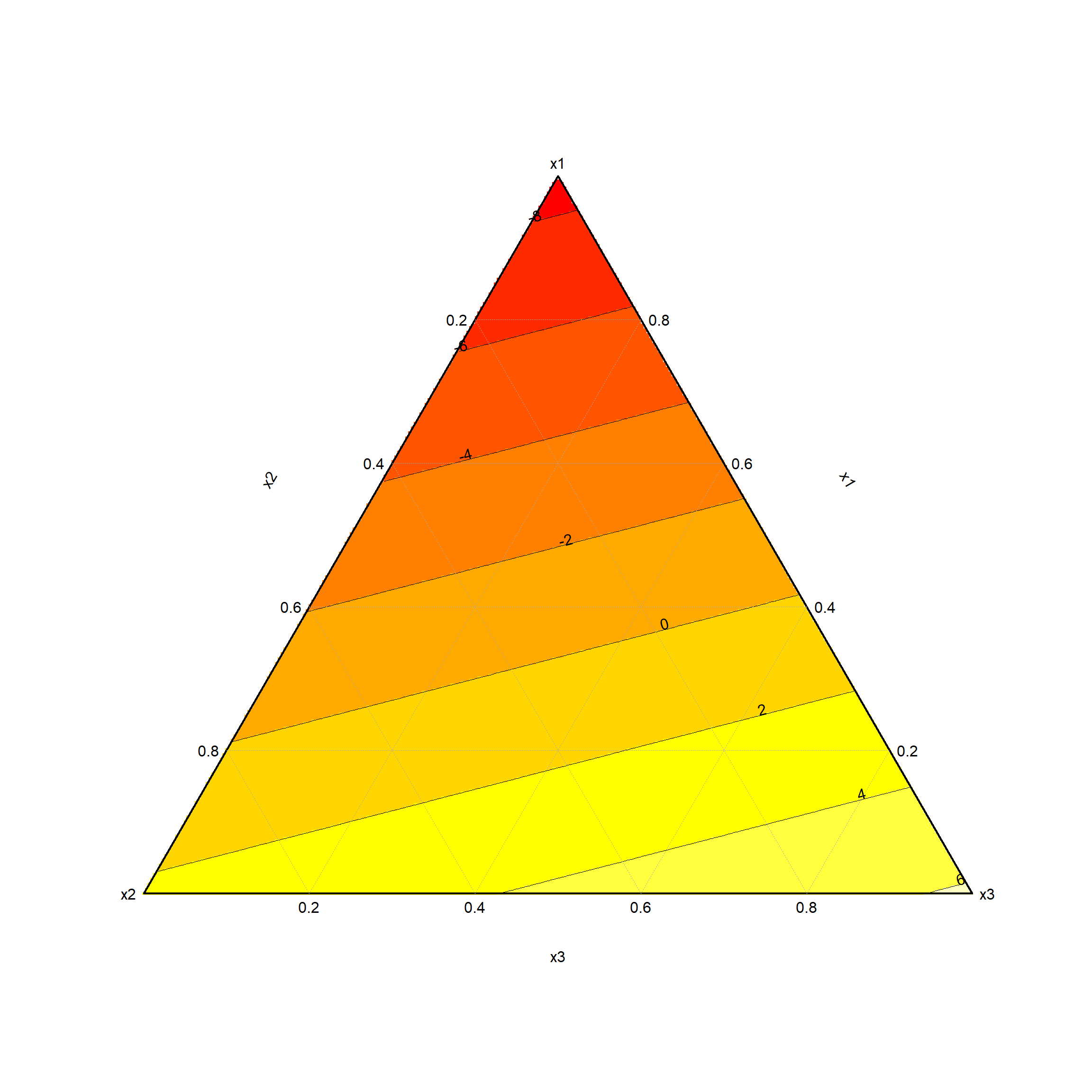
5.1.1 Método 1: Imputación por la media global
Código
abserror11 <- matrix(NA, ncol = 6, nrow = n)
errorcua11 <- matrix(NA, ncol = 6, nrow = n)
for (i in 1:6) {
tab2 <- resultados_df[-i, -(1:3)]
vr <- as.numeric(resultados_df[i, -(1:3)]) # valor real (vector)
ve <- as.numeric(apply(tab2, 2, mean)) # valor estimado (vector)
abserror11[,i] <- abs(vr - ve)
errorcua11[,i] <- (vr - ve)^2
resumenli[1, i+2] <- round(mean(abserror11[,i]),2)
resumenli[2, i+2] <- round(sqrt(mean(errorcua11[,i])),2)
}
colnames(abserror11) <- paste0("T", 1:6)5.1.2 Método 2: Imputación por media parcial
Código
abserror21 <- matrix(NA, ncol = 6, nrow = n)
errorcua21 <- matrix(NA, ncol = 6, nrow = n)
for (i in 1:6) {
vr <- as.numeric(resultados_df[i,-(1:3)]) # valor real
simg1 <- c(1,3,5) #Similares grupo 1
simg2 <- c(2,4,6) #Similares grupo 2
grupo <- if (i %in% simg1) simg1 else simg2
grupo_sin_sel <- grupo[grupo != i]
# Calcular promedio por columnas para las filas de ese grupo
ve<- colMeans(resultados_df[grupo_sin_sel, -c(1:3)]) # valor estimado
abserror21[,i] <- (abs(vr - ve))
errorcua21[,i] <- (vr - ve)^2
resumenli[3, i+2] <- round(mean(abserror21[,i]),2)
resumenli[4, i+2] <- round(sqrt(mean(errorcua21[,i])),2)
}
colnames(abserror21) <- paste0("T", 1:6)5.1.3 Método 3: Imputación por vecinos más cercanos (KNN)
Los vecinos más cercanos corresponden a aquellos tratamientos que, en la columna de clasificación de la tabla Tabla 12.2, presentan el valor igual a 1, es decir, a los tratamintos que presentaron menor distancia euclideana entre ellos.
Código
abserror31 <- matrix(NA, ncol = 6, nrow = n)
errorcua31 <- matrix(NA, ncol = 6, nrow = n)
for (i in 1:6) {
vr <- as.numeric(resultados_df[i,-(1:3)]) # valor real
vec <- tab_pesos %>%
filter(punto_inicial == i, clasificacion == 1) %>%
pull(punto_final)
# Calcular promedio por columnas para las filas de ese grupo
ve<- colMeans(resultados_df[vec, -c(1:3)]) # valor estimado
abserror31[,i] <- (abs(vr - ve))
errorcua31[,i] <- (vr - ve)^2
resumenli[5, i+2] <- round(mean(abserror31[,i]),2)
resumenli[6, i+2] <- round(sqrt(mean(errorcua31[,i])),2)
}
colnames(abserror31) <- paste0("T", 1:6)5.1.4 Método 4A: Modelo de regresion lineal canónico de Scheffé
Código
abserror411 <- matrix(NA, ncol = 6, nrow = n)
errorcua411 <- matrix(NA, ncol = 6, nrow = n)
for (i in 1:6) {
vr <- as.numeric(resultados_df[i,-(1:3)]) # valor real
ve <- numeric(n) # valor predicho
des_dfp <- resultados_df[-i,]
mod <- vector("list", n) # lista para guardar los modelos
# definición de los 7 tratamientos del simplex
tratamientos <- list(
c(1, 0, 0),
c(0.5, 0.5, 0),
c(0, 1, 0),
c(0, 0.5, 0.5),
c(0, 0, 1),
c(0.5, 0, 0.5),
c(1/3, 1/3, 1/3, 1/3, 1/3, 1/3, 1/3, 1/3, 1/3)
)
for (j in 1:n) {
# construir fórmula dinámica: yi ~ -1 + x1 + x2 + x3
f <- as.formula(paste0("y", j, " ~ -1 + x1 + x2 + x3"))
# ajustar modelo
mod[[j]] <- lm(f, data = des_dfp)
# armar newdata según tratamiento_sel
newdata <- data.frame(matrix(tratamientos[[i]], nrow = 1))
colnames(newdata) <- c("x1","x2","x3")
# predecir
ve[j] <- round(predict(mod[[j]], newdata = newdata),2)
}
abserror411[,i] <- (abs(vr - ve))
errorcua411[,i] <- (vr - ve)^2
resumenli[7, i+2] <- round(mean(abserror411[,i]),2)
resumenli[8, i+2] <- round(sqrt(mean(errorcua411[,i])),2)
}
colnames(abserror411) <- paste0("T", 1:6)5.1.5 Método 4B: Modelo de regresion cuadrático canónico de Scheffé
Código
abserror421 <- matrix(NA, ncol = 6, nrow = n)
errorcua421 <- matrix(NA, ncol = 6, nrow = n)
mod <- vector("list", n)
coefs <- matrix(NA, nrow = n, ncol = 6)
colnames(coefs) <- c("x1c","x2c","x3c",
"x1c_x2c", "x1c_x3c", "x2c_x3c")
for (i in 1:6) {
vr <- as.numeric(resultados_df[i,-(1:3)]) # valor real a estimar
des_dfp <- resultados_df[-i,] # tabla con valores disponibles
ve <- numeric(n) # vector para guardar predicciones
# definición de los 7 tratamientos del simplex
tratamientos <- list(
c(1, 0, 0),
c(0.5, 0.5, 0),
c(0, 1, 0),
c(0, 0.5, 0.5),
c(0, 0, 1),
c(0.5, 0, 0.5),
c(1/3, 1/3, 1/3, 1/3, 1/3, 1/3, 1/3, 1/3, 1/3))
for (j in 1:n) {
# fórmula cuadrática dinámica:
#yi ~ x1 + x2 + I(x1^2) + I(x2^2) + I(x1*x2)
f <- as.formula(paste0("y", j,
" ~ x1 + x2 + I(x1^2) + I(x2^2) + I(x1*x2)"))
# ajustar modelo
mod[[j]] <- lm(f, data = des_dfp)# modelo sin el valor a estimar
# obtener coeficientes transformados
a <- coefficients(mod[[j]])
# Paso a canónicos (siguiendo tu función mlmc)
a0 <- a["(Intercept)"]
a1 <- a["x1"]
a2 <- a["x2"]
a11 <- a["I(x1^2)"]
a22 <- a["I(x2^2)"]
a12 <- a["I(x1 * x2)"]
## Transformación a coeficientes canónicos ---
x1c <- a0 + a1 + a11
x2c <- a0 + a2 + a22
x3c <- a0
x1c_x2c <- a12 - a11 - a22
x1c_x3c <- -a11
x2c_x3c <- -a22
coefs[j, ] <- c(x1c, x2c, x3c, x1c_x2c, x1c_x3c, x2c_x3c)
# armar newdata según tratamiento sel
newdata <- data.frame(matrix(tratamientos[[i]], nrow = 1))
x1 <- newdata[,1]; x2 <- newdata[,2]; x3 <- newdata[,3]
# predecir
ve[j] <- coefs[j,"x1c"]*x1 + coefs[j,"x2c"]*x2 + coefs[j,"x3c"]*x3 +
coefs[j,"x1c_x2c"]*x1*x2 + coefs[j,"x1c_x3c"]*x1*x3 +
coefs[j,"x2c_x3c"]*x2*x3}
abserror421[,i] <- (abs(vr - ve))
errorcua421[,i] <- (vr - ve)^2
resumenli[9, i+2] <- round(mean(abserror421[,i]),2)
resumenli[10, i+2] <- round(sqrt(mean(errorcua421[,i])),2)
}5.2 Modelo cuadrático
Se emplea la misma función descrita anteriormente, modificando el argumento a mod = 2, así:
Código
[1] 1.8662513 0.9004555 0.2291747 1.1170002 1.7076118 1.4702207 1.1167179
[8] 1.1241400 1.4809150Se simulan 1000 modelos generados a partir de un modelo cuadrático canónico de Scheffé. A continuación, se presenta uno de ellos:
coefficients Std.err t.value Prob
x1 3.4917656 1.104263 3.1620773 0.050789711
x2 -7.1187491 1.103061 -6.4536290 0.007546374
x3 -3.1815329 1.103061 -2.8842754 0.063307769
x2:x1 1.4273828 4.846615 0.2945113 0.787570499
x3:x1 -0.7669499 4.846615 -0.1582445 0.884316180
x2:x3 19.6271745 4.834877 4.0594980 0.026944830
Residual standard error: 1.109706 on 3 degrees of freedom
Corrected Multiple R-squared: 0.9477758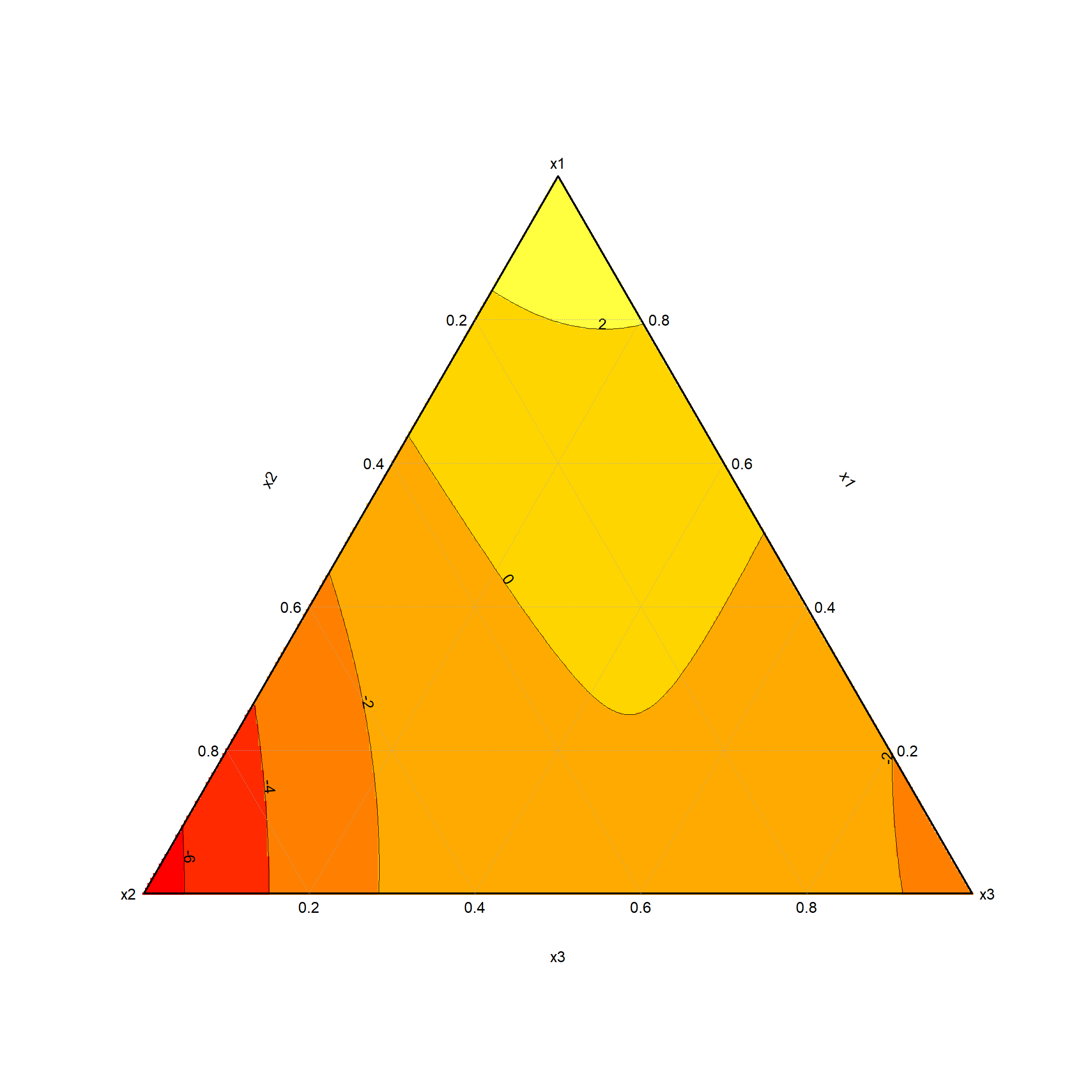
La imputación se lleva a cabo para cada uno de los métodos empleando los mismos procedimientos y códigos utilizados previamente en el modelo lineal.
6 Resultados
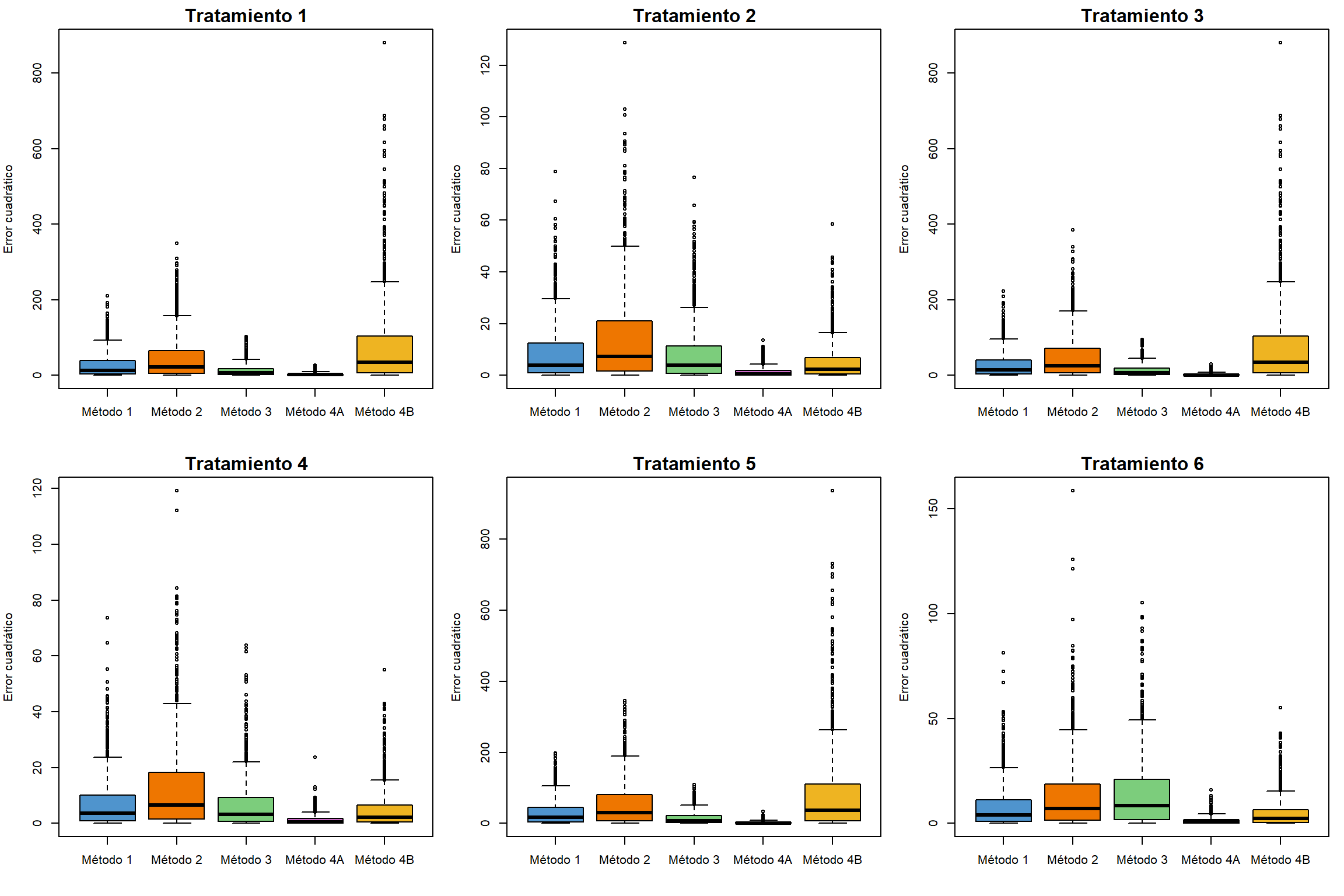
6.1 Modelo lineal
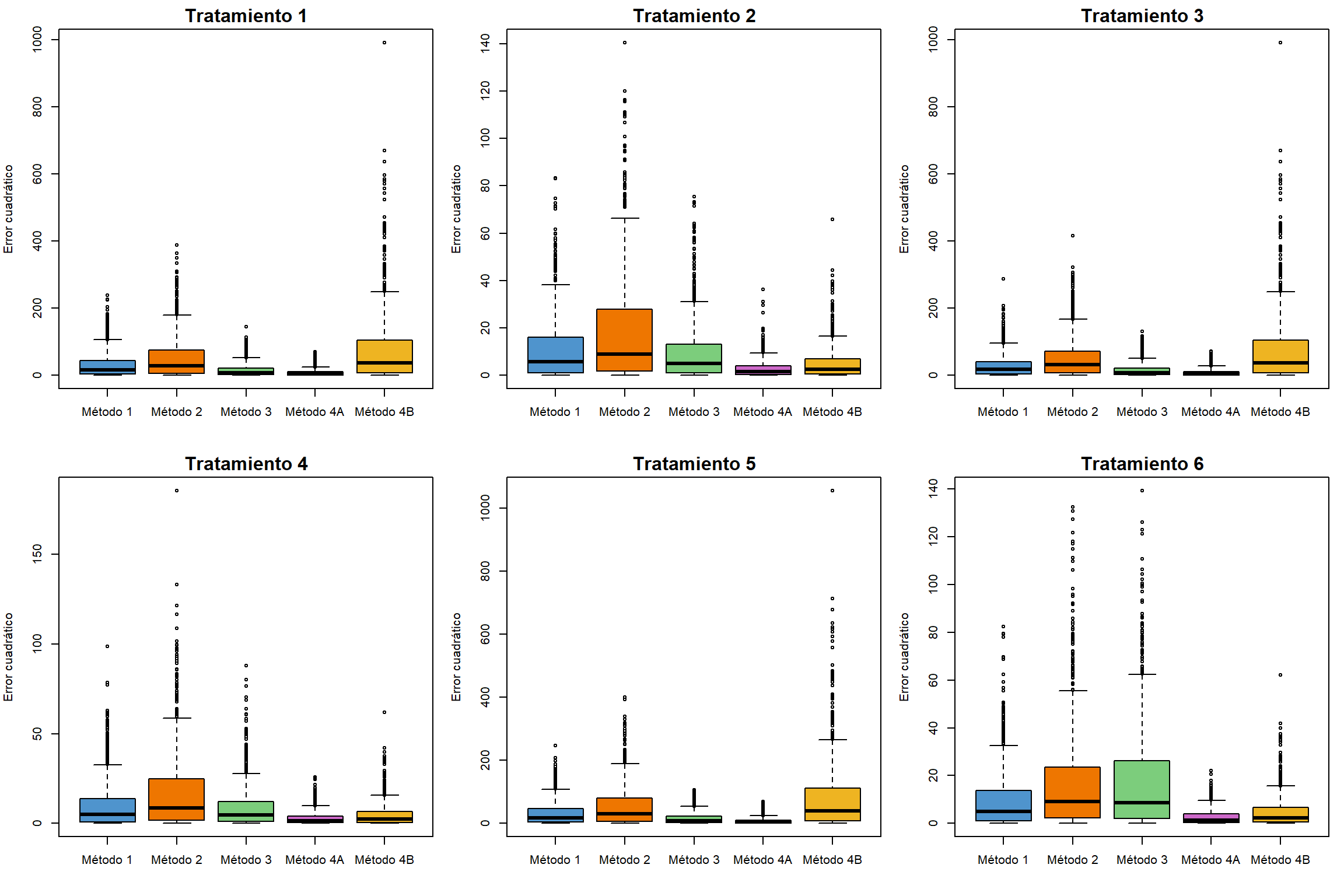
Método de imputación | Métricas de error | T 1 | T 2 | T 3 | T 4 | T 5 | T 6 |
|---|---|---|---|---|---|---|---|
Método 1 | EAM | 4.20 | 2.37 | 4.35 | 2.21 | 4.56 | 2.27 |
RECM | 5.21 | 2.92 | 5.33 | 2.73 | 5.54 | 2.81 | |
Método 2 | EAM | 5.55 | 3.11 | 5.80 | 2.91 | 6.09 | 3.00 |
RECM | 6.89 | 3.80 | 7.07 | 3.59 | 7.38 | 3.70 | |
Método 3 | EAM | 2.92 | 2.28 | 2.99 | 2.13 | 3.14 | 3.19 |
RECM | 3.63 | 2.85 | 3.67 | 2.64 | 3.85 | 3.86 | |
Método 4 - I | EAM | 1.37 | 0.92 | 1.29 | 0.86 | 1.33 | 0.94 |
RECM | 1.71 | 1.17 | 1.62 | 1.10 | 1.64 | 1.18 | |
Método 4 - II | EAM | 7.03 | 1.81 | 7.03 | 1.76 | 7.25 | 1.76 |
RECM | 8.84 | 2.28 | 8.84 | 2.21 | 9.11 | 2.21 |
6.2 Modelo cuadrático
Método de imputación | Métricas de error | T 1 | T 2 | T 3 | T 4 | T 5 | T 6 |
|---|---|---|---|---|---|---|---|
Método 1 | EAM | 4.54 | 2.66 | 4.53 | 2.54 | 4.64 | 2.57 |
RECM | 5.58 | 3.29 | 5.47 | 3.16 | 5.62 | 3.15 | |
Método 2 | EAM | 6.00 | 3.48 | 6.00 | 3.36 | 6.09 | 3.41 |
RECM | 7.33 | 4.28 | 7.21 | 4.15 | 7.39 | 4.16 | |
Método 3 | EAM | 3.18 | 2.55 | 3.18 | 2.43 | 3.26 | 3.43 |
RECM | 3.95 | 3.13 | 3.88 | 3.00 | 3.97 | 4.20 | |
Método 4 - I | EAM | 2.19 | 1.43 | 2.34 | 1.37 | 2.25 | 1.34 |
RECM | 2.70 | 1.75 | 2.88 | 1.71 | 2.77 | 1.63 | |
Método 4 - II | EAM | 7.07 | 1.82 | 7.07 | 1.77 | 7.29 | 1.77 |
RECM | 8.82 | 2.27 | 8.82 | 2.20 | 9.09 | 2.20 |
7 Anexos
Tratamiento inicial | Tratamiento final | Distancia | Clasificación |
|---|---|---|---|
1 | 2 | 0.50 | 1 |
1 | 6 | 0.50 | 1 |
1 | 7 | 0.67 | 2 |
1 | 4 | 0.86 | 2 |
1 | 3 | 1.00 | 3 |
1 | 5 | 1.00 | 3 |
2 | 7 | 0.33 | 1 |
2 | 1 | 0.50 | 2 |
2 | 3 | 0.50 | 2 |
2 | 4 | 0.50 | 2 |
2 | 6 | 0.50 | 2 |
2 | 5 | 0.86 | 3 |
3 | 2 | 0.50 | 1 |
3 | 4 | 0.50 | 1 |
3 | 7 | 0.67 | 2 |
3 | 6 | 0.86 | 2 |
3 | 1 | 1.00 | 3 |
3 | 5 | 1.00 | 3 |
4 | 7 | 0.33 | 1 |
4 | 2 | 0.50 | 2 |
4 | 3 | 0.50 | 2 |
4 | 5 | 0.50 | 2 |
4 | 6 | 0.50 | 2 |
4 | 1 | 0.86 | 3 |
5 | 4 | 0.50 | 1 |
5 | 6 | 0.50 | 1 |
5 | 7 | 0.67 | 2 |
5 | 2 | 0.86 | 2 |
5 | 1 | 1.00 | 3 |
5 | 3 | 1.00 | 3 |
6 | 1 | 0.50 | 1 |
6 | 2 | 0.50 | 1 |
6 | 7 | 0.67 | 2 |
6 | 4 | 0.86 | 2 |
6 | 3 | 1.00 | 3 |
6 | 5 | 1.00 | 3 |
7 | 2 | 0.33 | 1 |
7 | 4 | 0.33 | 1 |
7 | 6 | 0.33 | 1 |
7 | 1 | 0.67 | 2 |
7 | 3 | 0.67 | 2 |
7 | 5 | 0.67 | 2 |