Librerías requeridas: dplyr, flextable, car, nortest4 Replicación en R de caso de estudio: Charoo et al. (2012)
Quality by design approach for formulation development: a case study of dispersible tablets, artículo escrito por Charoo et al. (2012)
5 Artículo de estudio
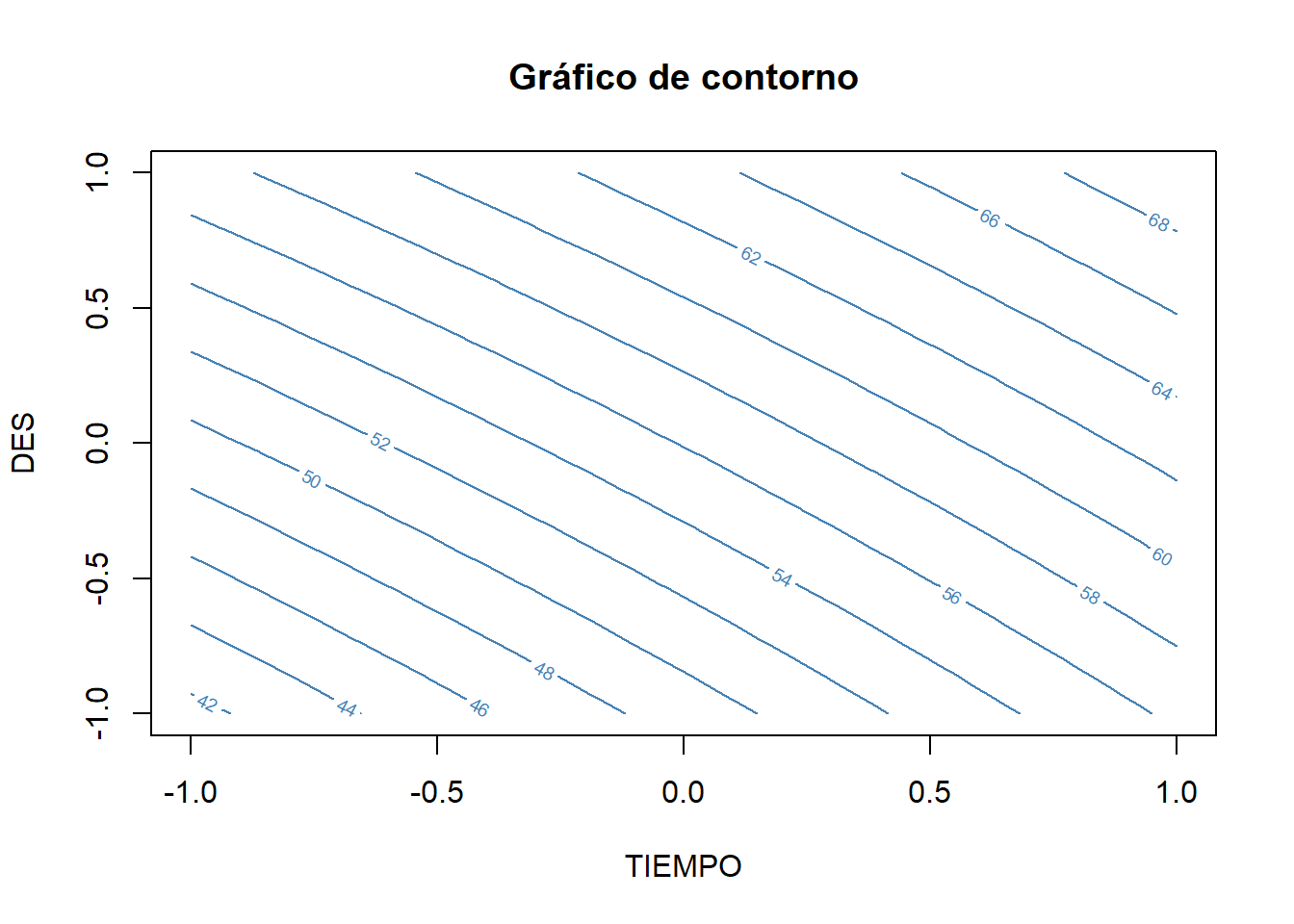
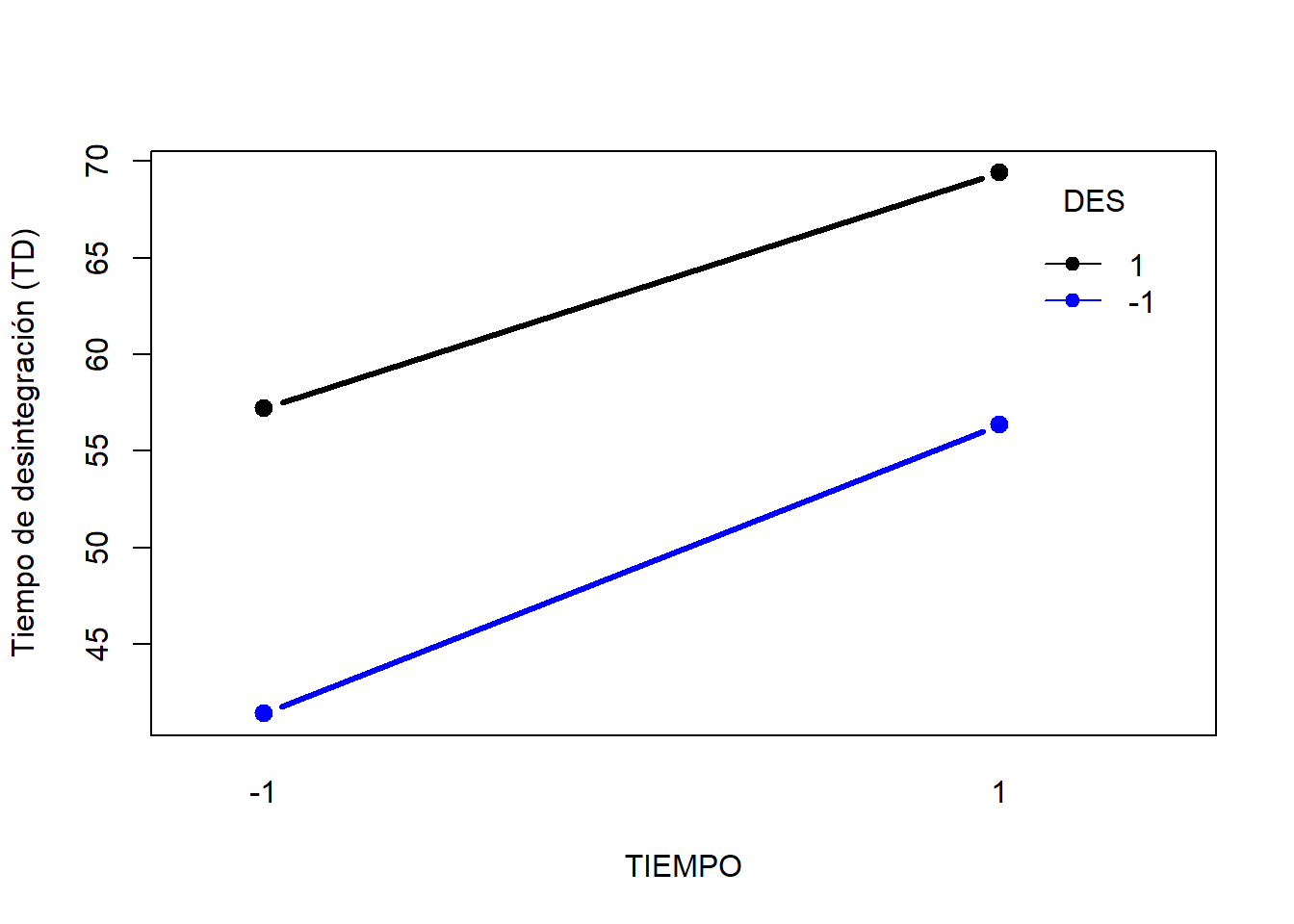
En el artículo publicado por Charoo et al. (2012), “Quality by design approach for formulation development: a case study of dispersible tablets”, se describen varios diseños experimentales relacionados con la fabricación de tabletas orodispersables de diclofenaco. A continuación, se muestra una réplica del Estudio No. 7 descrito en el artículo de referencia, orientado a evaluar la influencia de la proporción de desintegrante (DES) y el tiempo de mezcla (TIEMPO) sobre el tiempo de desintegración (TD).
Véase “Guía para la implementación de diseños experimentales en la industria farmacéutica” para una explicación detallada de los cálculos presentados.
6 Simulación de respuestas
El artículo de Charoo et al. (2012) reporta únicamente el valor promedio de las tres réplicas junto con su desviación estándar, por lo que se requiere simular los datos individuales. Para este propósito, se construyó la función simuesblo, disponible en el archivo funciones.R del repositorio ncortes/Disexp_Industria_Farmaceutica en GitHub (funcionesR?).
Los valores simulados para el tiempo de desintegración son: 41.16, 58.34, 55.19, 70.69, 41.65, 56.11, 57.57, 68.1
7 Construcción del diseño experimental
TRAT | REP | TIEMPO | DES | TD |
|---|---|---|---|---|
T1 | R1 | -1 | -1 | 41.16 |
T2 | R1 | -1 | 1 | 58.34 |
T3 | R1 | 1 | -1 | 55.19 |
T4 | R1 | 1 | 1 | 70.69 |
T1 | R2 | -1 | -1 | 41.65 |
T2 | R2 | -1 | 1 | 56.11 |
T3 | R2 | 1 | -1 | 57.57 |
T4 | R2 | 1 | 1 | 68.10 |
8 Modelo de regresión
Call:
lm(formula = TD ~ TIEMPO * DES, data = dis)
Residuals:
1 2 3 4 5 6 7 8
-0.245 1.115 -1.190 1.295 0.245 -1.115 1.190 -1.295
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 56.1012 0.5242 107.025 4.57e-08 ***
TIEMPO 6.7863 0.5242 12.946 0.000205 ***
DES 7.2088 0.5242 13.752 0.000162 ***
TIEMPO:DES -0.7013 0.5242 -1.338 0.251961
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.483 on 4 degrees of freedom
Multiple R-squared: 0.989, Adjusted R-squared: 0.9807
F-statistic: 119.5 on 3 and 4 DF, p-value: 0.00022749 ANOVA
Analysis of Variance Table
Response: TD
Df Sum Sq Mean Sq F value Pr(>F)
TIEMPO 1 368.43 368.43 167.6042 0.0002054 ***
DES 1 415.73 415.73 189.1234 0.0001620 ***
TIEMPO:DES 1 3.93 3.93 1.7897 0.2519609
Residuals 4 8.79 2.20
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 110 Verificación de supuestos
Código
# Residuos del modelo
residuales <- mod$residuals
# Prueba de homogeneidad de varianzas
bt <- bartlett.test(residuales~TRAT, data=dis)
# Prueba de normalidad de Kolmogorov-Smirnov
ks <- ks.test(residuales,"pnorm",mean(residuales), sd(residuales))
# Prueba de normalidad de Shapiro-Wilk
sp <- shapiro.test(residuales)Pruebas | Estadístico | p-valor | Interpretación |
|---|---|---|---|
Prueba de bartlett | 1.4882 | 0.6850 | Los residuales son homocedásticos |
Kolmogorov-Smirnov | 0.2151 | 0.7823 | Los residuales son normales |
Shapiro-Wilk | 0.8526 | 0.1013 | Los residuales son normales |
11 Graficos de interacción
Código
12 Gráfico de superficie
Código
# Definir la malla
x1 <- seq(min(dis$TIEMPO), max(dis$TIEMPO), length = 30)
x2 <- seq(min(dis$DES), max(dis$DES), length = 30)
# Crear cuadrícula
grid <- expand.grid(TIEMPO = x1, DES = x2)
# Predecir con el modelo en cada punto de la malla
grid$z <- predict(mod, newdata = grid)
# Pasar a matriz para persp/contour
z <- matrix(grid$z, nrow = length(x1), ncol = length(x2))
# --- Gráfico de superficie ---
persp(x1, x2, z,
theta = 30, phi = 25,
col = NA,
border = "steelblue",
lwd = 1,
ticktype = "detailed",
xlab = "TIEMPO", ylab = "DES", zlab = "TD",
cex.lab = 0.8,
cex.axis = 0.8,
cex.main = 0.8,
main = "Gráfico de superficie de respuesta")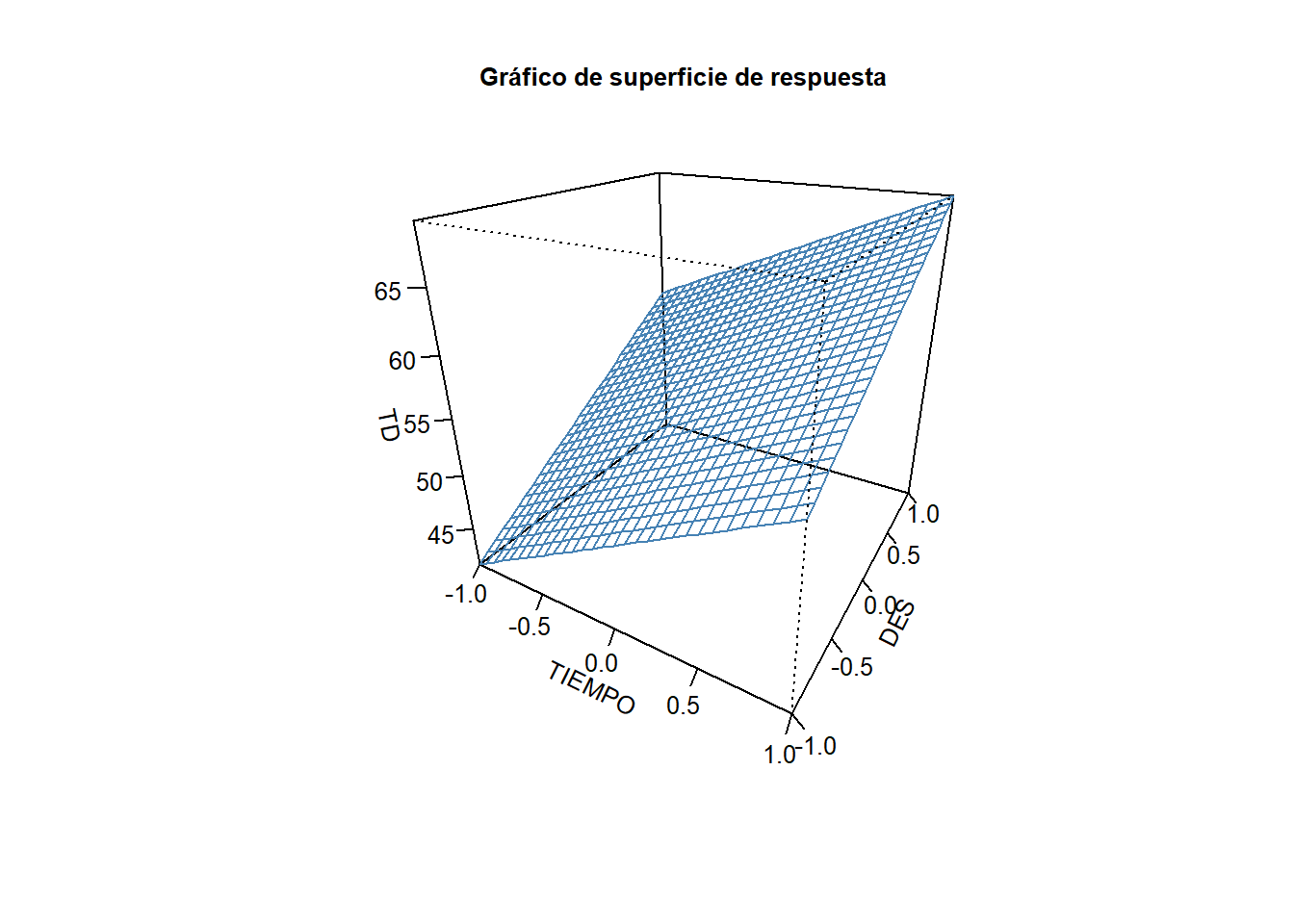
13 Gráfico de contorno
Código