Librerías requeridas: FrF2, dplyr, knitr, flextable, ggplot2, gridExtra8 Replicación en R de caso de estudio: Rahamathulla et al. (2022)
2³ factorial design and optimization of effervescent floating matrix tablet of neratinib
9 Artículo de estudio
Rahamathulla et al. (2022) presenta en su artículo “2³ factorial design and optimization of effervescent floating matrix tablet of neratinib”, la formulación de una tableta con sistema de liberación flotante de neratinib, empleada en el tratamiento del cáncer de mama. Este fármaco presenta una baja solubilidad en agua a pH elevado, lo que limita su eficacia terapéutica y, en consecuencia, su biodisponibilidad. Por esta razón, es preferible que la absorción del fármaco se produzca en el estómago, donde el pH es bajo. Sin embargo, la permanencia del fármaco en el estómago se ve afectada por factores como el vaciado y la motilidad gástrica, entre otros. Para superar esta limitación, se diseñó una tableta con un sistema de liberación flotante, con el objetivo de prolongar el tiempo de residencia gástrica y, de este modo, incrementar su biodisponibilidad.
Para el desarrollo de la matriz se empleó un diseño factorial 2³ con puntos centrales, en el cual se evaluaron como factores las concentraciones de los excipientes hidroxipropilmetilcelulosa, carbopol 940 y celulosa microcristalina. El artículo presenta la evaluación de varias variables respuesta; sin embargo, en este ejercicio solo se tendrá en cuenta el índice de Carr (CARR) y la friabilidad (FRIAB).
Véase “Guía para la implementación de diseños experimentales en la industria farmacéutica” para una explicación detallada de los cálculos presentados.
10 Factores evaluados
Variables | Nivel (-1) | Nivel (0) | Nivel (1) |
|---|---|---|---|
Hidroxipropilmetilcelulosa (HPMC) | 25 mg | 37.5 mg | 50 mg |
Carbopol 940 (CARB) | 25 mg | 37.5 mg | 50 mg |
Celulosa microcristalina (MCC) | 15 mg | 20 mg | 25 mg |
11 Simulación de respuestas
El artículo de (Rahamathulla et al. 2022) reporta únicamente el valor promedio de las tres réplicas junto con su desviación estándar, , por lo que se requiere simular los datos individuales. Para este propósito, se construyó la función simuesblo, disponible en el archivo funciones.R del repositorio ncortes/Disexp_Industria_Farmaceutica en GitHub (funcionesR?).
Los valores simulados para el índice de Carr son: 12.2, 13.94, 14.76, 4.35, 11.61, 10.7, 6.93, 10.07, 2.36, 12.5, 14, 12.66, 7.84, 11.02, 8.75, 5.37, 9.59, 1.28, 14.13, 15.64, 13.36, 6.03, 9.66, 5.03, 4.58, 9.77, 3.85
12 Diseño factorial completo 2³ con puntos centrales
Código
# Diseño de variables codificadas
dis1_cod <- data.frame(
TRAT = rep(paste("TRAT", seq(1,9)),3),
HPMC = rep(c(1, -1, -1, 1, 1, -1, 0, -1, 1),3),
CARB = rep(c(1, -1, -1, 1, -1, 1, 0, 1, -1),3),
MCC = rep(c(1, 1, -1, -1, -1, -1, 0, 1, 1),3),
CARR)
# Diseño de variables naturales
dis1_nat <- data.frame(
TRAT_ = rep(paste("TRAT", seq(1,9)),3),
HPMC_ = rep(c(50, 25, 25, 50, 50, 25, 37.5, 25, 50), 3),
CARB_ = rep(c(50, 25, 25, 50, 25, 50, 37.5, 50, 25), 3),
MCC_ = rep(c(25, 25, 15, 15, 15, 15, 20, 25, 25), 3),
CARR)TRATAMIENTOS | HPMC(c) | CARB(c) | MCC(c) | HPMC(n) | CARB(n) | MCC(n) | CARR |
|---|---|---|---|---|---|---|---|
TRAT 1 | 1 | 1 | 1 | 50.0 | 50.0 | 25 | 12.20 |
TRAT 2 | -1 | -1 | 1 | 25.0 | 25.0 | 25 | 13.94 |
TRAT 3 | -1 | -1 | -1 | 25.0 | 25.0 | 15 | 14.76 |
TRAT 4 | 1 | 1 | -1 | 50.0 | 50.0 | 15 | 4.35 |
TRAT 5 | 1 | -1 | -1 | 50.0 | 25.0 | 15 | 11.61 |
TRAT 6 | -1 | 1 | -1 | 25.0 | 50.0 | 15 | 10.70 |
TRAT 7 | 0 | 0 | 0 | 37.5 | 37.5 | 20 | 6.93 |
TRAT 8 | -1 | 1 | 1 | 25.0 | 50.0 | 25 | 10.07 |
TRAT 9 | 1 | -1 | 1 | 50.0 | 25.0 | 25 | 2.36 |
TRAT 1 | 1 | 1 | 1 | 50.0 | 50.0 | 25 | 12.50 |
TRAT 2 | -1 | -1 | 1 | 25.0 | 25.0 | 25 | 14.00 |
TRAT 3 | -1 | -1 | -1 | 25.0 | 25.0 | 15 | 12.66 |
TRAT 4 | 1 | 1 | -1 | 50.0 | 50.0 | 15 | 7.84 |
TRAT 5 | 1 | -1 | -1 | 50.0 | 25.0 | 15 | 11.02 |
TRAT 6 | -1 | 1 | -1 | 25.0 | 50.0 | 15 | 8.75 |
TRAT 7 | 0 | 0 | 0 | 37.5 | 37.5 | 20 | 5.37 |
TRAT 8 | -1 | 1 | 1 | 25.0 | 50.0 | 25 | 9.59 |
TRAT 9 | 1 | -1 | 1 | 50.0 | 25.0 | 25 | 1.28 |
TRAT 1 | 1 | 1 | 1 | 50.0 | 50.0 | 25 | 14.13 |
TRAT 2 | -1 | -1 | 1 | 25.0 | 25.0 | 25 | 15.64 |
TRAT 3 | -1 | -1 | -1 | 25.0 | 25.0 | 15 | 13.36 |
TRAT 4 | 1 | 1 | -1 | 50.0 | 50.0 | 15 | 6.03 |
TRAT 5 | 1 | -1 | -1 | 50.0 | 25.0 | 15 | 9.66 |
TRAT 6 | -1 | 1 | -1 | 25.0 | 50.0 | 15 | 5.03 |
TRAT 7 | 0 | 0 | 0 | 37.5 | 37.5 | 20 | 4.58 |
TRAT 8 | -1 | 1 | 1 | 25.0 | 50.0 | 25 | 9.77 |
TRAT 9 | 1 | -1 | 1 | 50.0 | 25.0 | 25 | 3.85 |
(c): notación codificada; (n): notación natural | |||||||
12.1 Indice de Carr
12.1.1 Modelo de regresión
Call:
lm.default(formula = CARR ~ HPMC * CARB * MCC, data = dis1_cod)
Residuals:
Min 1Q Median 3Q Max
-4.7526 -0.5551 0.2432 1.1816 3.0032
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 9.3326 0.4038 23.111 2.27e-15 ***
HPMC -1.7267 0.4283 -4.031 0.000713 ***
CARB -0.5492 0.4283 -1.282 0.215211
MCC 0.1483 0.4283 0.346 0.732909
HPMC:CARB 1.9883 0.4283 4.642 0.000178 ***
HPMC:MCC -0.4975 0.4283 -1.162 0.259811
CARB:MCC 1.9817 0.4283 4.627 0.000184 ***
HPMC:CARB:MCC 1.8025 0.4283 4.208 0.000476 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 2.098 on 19 degrees of freedom
Multiple R-squared: 0.8081, Adjusted R-squared: 0.7375
F-statistic: 11.43 on 7 and 19 DF, p-value: 1.219e-0512.1.2 Verificación de supuestos
Código
# Residuos del modelo
residuales <- mod1$residuals
# Prueba de homogeneidad de varianzas
modx <- lm(CARR~TRAT, dis1_cod)
residualesx <- residuals(modx)
bt <- bartlett.test(residualesx~TRAT, data=dis1_cod)
# Prueba de normalidad de Kolmogorov-Smirnov
ks <- ks.test(residuales,"pnorm",mean(residuales), sd(residuales))
# Prueba de normalidad de Shapiro-Wilk
sp <- shapiro.test(residuales)Pruebas | p-valor | Interpretación |
|---|---|---|
Prueba de bartlett | 0.3681 | Los residuales son homocedásticos |
Kolmogorov-Smirnov | 0.5120 | Los residuales son normales |
Shapiro-Wilk | 0.0501 | Los residuales son normales |
Analysis of Variance Table
Response: CARR
Df Sum Sq Mean Sq F value Pr(>F)
HPMC 1 71.553 71.553 16.2515 0.0007133 ***
CARB 1 7.238 7.238 1.6439 0.2152110
MCC 1 0.528 0.528 0.1199 0.7329086
HPMC:CARB 1 94.883 94.883 21.5503 0.0001777 ***
HPMC:MCC 1 5.940 5.940 1.3492 0.2598107
CARB:MCC 1 94.248 94.248 21.4061 0.0001840 ***
HPMC:CARB:MCC 1 77.976 77.976 17.7103 0.0004761 ***
Residuals 19 83.655 4.403
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 112.1.3 Graficos de interacción
Código
#jpeg("mi_grafico.jpg", width = 1400, height = 800, res = 150)
dis1_cod2 <- subset(dis1_cod, CARB %in% c(-1, 1))
dis1_cod2$CARB <- factor(dis1_cod2$CARB)
p1 <- ggplot(dis1_cod2, aes(x = HPMC, y = CARR, group = CARB, color = CARB)) +
stat_summary(fun = mean, geom = "line", linewidth = 1.2) +
stat_summary(fun = mean, geom = "point", size = 3) +
scale_x_continuous(breaks = c(-1, 1)) +
scale_color_manual(values = c("blue", "black")) +
labs(x = "HPMC", y = "CARR", color = "CARB") +
theme_minimal()
dis1_cod2 <- subset(dis1_cod, CARB %in% c(-1, 1))
dis1_cod2$CARB <- factor(dis1_cod2$CARB)
p2 <- ggplot(dis1_cod2, aes(x = MCC, y = CARR, group = CARB, color = CARB)) +
stat_summary(fun = mean, geom = "line", linewidth = 1.2) +
stat_summary(fun = mean, geom = "point", size = 3) +
scale_x_continuous(breaks = c(-1, 1)) +
scale_color_manual(values = c("blue", "black")) +
labs(x = "MCC", y = "CARR", color = "CARB") +
theme_minimal()
dis1_cod2 <- subset(dis1_cod, MCC %in% c(-1, 1))
dis1_cod2$MCC <- factor(dis1_cod2$MCC)
p3 <- ggplot(dis1_cod2, aes(x = HPMC, y = CARR, group = MCC, color = MCC)) +
stat_summary(fun = mean, geom = "line", linewidth = 1.2) +
stat_summary(fun = mean, geom = "point", size = 3) +
scale_x_continuous(breaks = c(-1, 1)) +
scale_color_manual(values = c("blue", "black")) +
labs(x = "HPMC", y = "CARR", color = "MCC") +
theme_minimal()
grid.arrange(p1, p2, p3, ncol = 3)
#dev.off()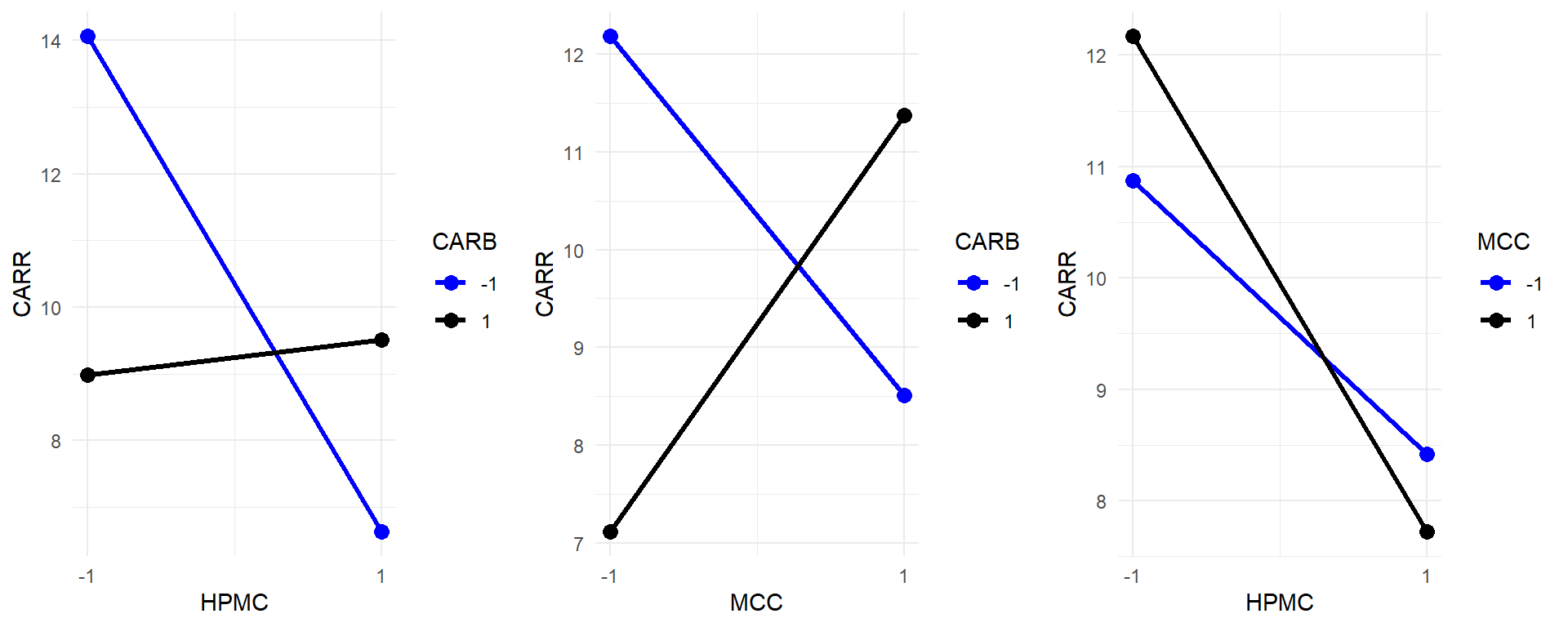
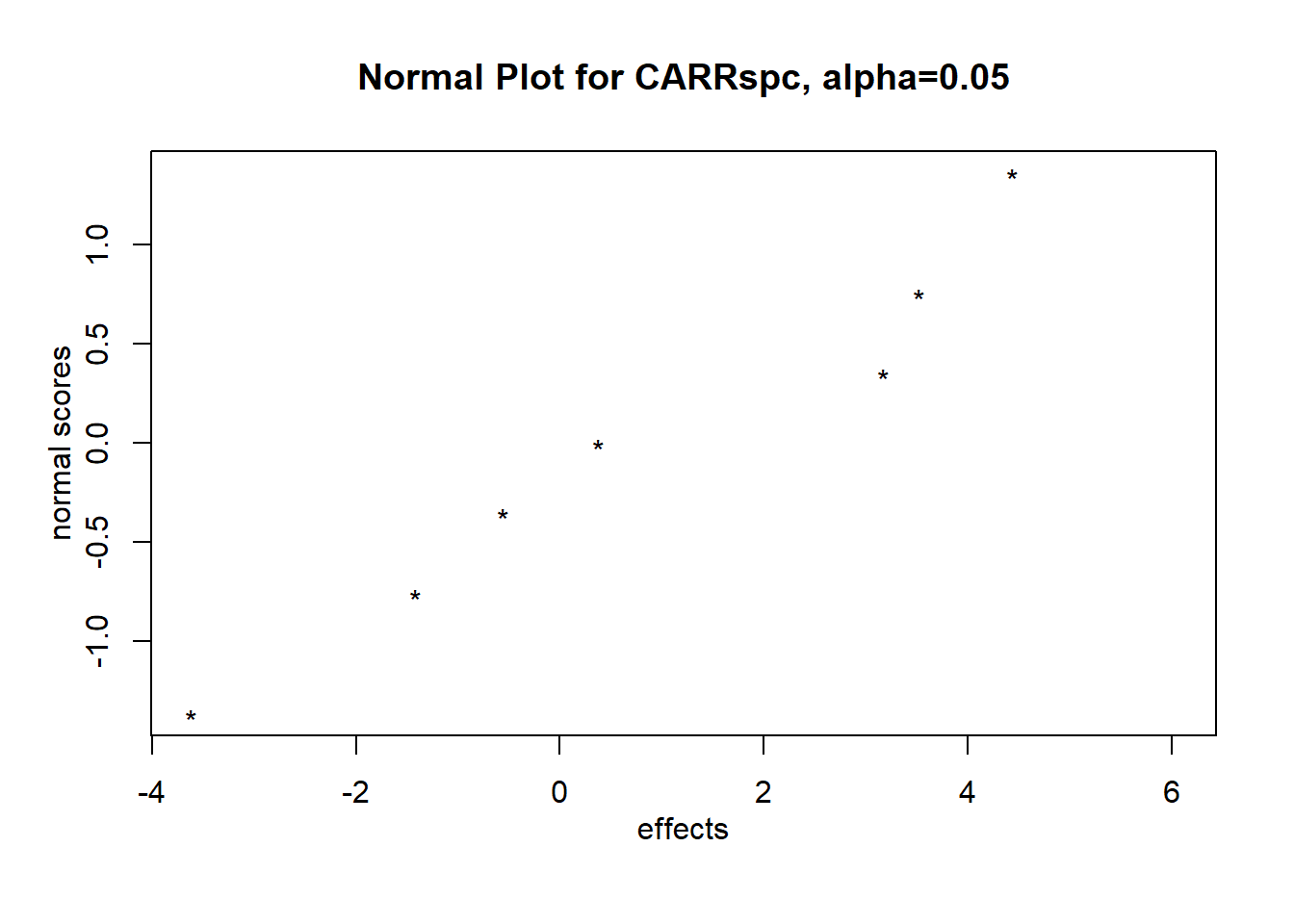
13 Diseño factorial completo 2³ no replicado y sin puntos centrales
Para construir un diseño factorial completo 2³ no replicado y sin puntos centrales, se eliminan las filas correspondientes al punto central (TRAT 7) y se incorpora directamente la variable respuesta CARR, tal como fue reportada en el artículo, sin realizar simulación de réplicas.
Código
# Diseño de variables codificadas
CARRspc <- carr_mean[-7]
dis2_cod <- data.frame(
TRAT = paste("TRAT", seq(1,8)),
HPMC = c(1, -1, -1, 1, 1, -1, -1, 1),
CARB = c(1, -1, -1, 1, -1, 1, 1, -1),
MCC = c(1, 1, -1, -1, -1, -1, 1, 1),
CARRspc)
# Diseño de variables naturales
dis2_nat <- data.frame(
TRAT = paste("TRAT", seq(1,8)),
HPMC_ = c(50, 25, 25, 50, 50, 25, 25, 50),
CARB_ = c(50, 25, 25, 50, 25, 50, 50, 25),
MCC_ = c(25, 25, 15, 15, 15, 15, 25, 25),
CARRspc)TRATAMIENTOS | HPMC(c) | CARB(c) | MCC(c) | HPMC(n) | CARB(n) | MCC(n) | CARR |
|---|---|---|---|---|---|---|---|
TRAT 1 | 1 | 1 | 1 | 50 | 50 | 25 | 12.71 |
TRAT 2 | -1 | -1 | 1 | 25 | 25 | 25 | 13.87 |
TRAT 3 | -1 | -1 | -1 | 25 | 25 | 15 | 14.20 |
TRAT 4 | 1 | 1 | -1 | 50 | 50 | 15 | 5.28 |
TRAT 5 | 1 | -1 | -1 | 50 | 25 | 15 | 10.79 |
TRAT 6 | -1 | 1 | -1 | 25 | 50 | 15 | 8.00 |
TRAT 7 | -1 | 1 | 1 | 25 | 50 | 25 | 10.20 |
TRAT 8 | 1 | -1 | 1 | 50 | 25 | 25 | 3.00 |
(c): notación codificada; (n): notación natural | |||||||
14 Diseño factorial completo 2³ no replicado con puntos centrales
Para construir un diseño factorial completo 2³ no replicado con puntos centrales, se incorpora directamente la variable respuesta CARR, tal como fue reportada en el artículo, sin realizar simulación de réplicas.
Código
# Diseño de variables codificadas
CARRmean <- carr_mean
dis3_cod <- data.frame(
TRAT = paste("TRAT", seq(1,9)),
HPMC = c(1, -1, -1, 1, 1, -1, 0, -1, 1),
CARB = c(1, -1, -1, 1, -1, 1, 0, 1, -1),
MCC = c(1, 1, -1, -1, -1, -1, 0, 1, 1),
CARRmean)
# Diseño de variables naturales
dis3_nat <- data.frame(
TRAT = paste("TRAT", seq(1,9)),
HPMC_ = c(50, 25, 25, 50, 50, 25, 37.5, 25, 50),
CARB_ = c(50, 25, 25, 50, 25, 50, 37.5, 50, 25),
MCC_ = c(25, 25, 15, 15, 15, 15, 20, 25, 25),
CARRmean)TRATAMIENTOS | HPMC(c) | CARB(c) | MCC(c) | HPMC(n) | CARB(n) | MCC(n) | CARR |
|---|---|---|---|---|---|---|---|
TRAT 1 | 1 | 1 | 1 | 50.0 | 50.0 | 25 | 12.71 |
TRAT 2 | -1 | -1 | 1 | 25.0 | 25.0 | 25 | 13.87 |
TRAT 3 | -1 | -1 | -1 | 25.0 | 25.0 | 15 | 14.20 |
TRAT 4 | 1 | 1 | -1 | 50.0 | 50.0 | 15 | 5.28 |
TRAT 5 | 1 | -1 | -1 | 50.0 | 25.0 | 15 | 10.79 |
TRAT 6 | -1 | 1 | -1 | 25.0 | 50.0 | 15 | 8.00 |
TRAT 7 | 0 | 0 | 0 | 37.5 | 37.5 | 20 | 6.00 |
TRAT 8 | -1 | 1 | 1 | 25.0 | 50.0 | 25 | 10.20 |
TRAT 9 | 1 | -1 | 1 | 50.0 | 25.0 | 25 | 3.00 |
(c): notación codificada; (n): notación natural | |||||||
Call:
lm.default(formula = CARRmean ~ HPMC * CARB * MCC, data = dis3_cod)
Residuals:
1 2 3 4 5 6 7 8 9
0.4174 0.4174 0.4174 0.4174 0.4174 0.4174 -3.3389 0.4174 0.4174
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 9.3389 1.1805 7.911 0.080 .
HPMC -1.8112 1.2521 -1.447 0.385
CARB -0.7087 1.2521 -0.566 0.672
MCC 0.1888 1.2521 0.151 0.905
HPMC:CARB 1.7587 1.2521 1.405 0.394
HPMC:MCC -0.2787 1.2521 -0.223 0.861
CARB:MCC 2.2188 1.2521 1.772 0.327
HPMC:CARB:MCC 1.5863 1.2521 1.267 0.425
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 3.541 on 1 degrees of freedom
Multiple R-squared: 0.902, Adjusted R-squared: 0.216
F-statistic: 1.315 on 7 and 1 DF, p-value: 0.5879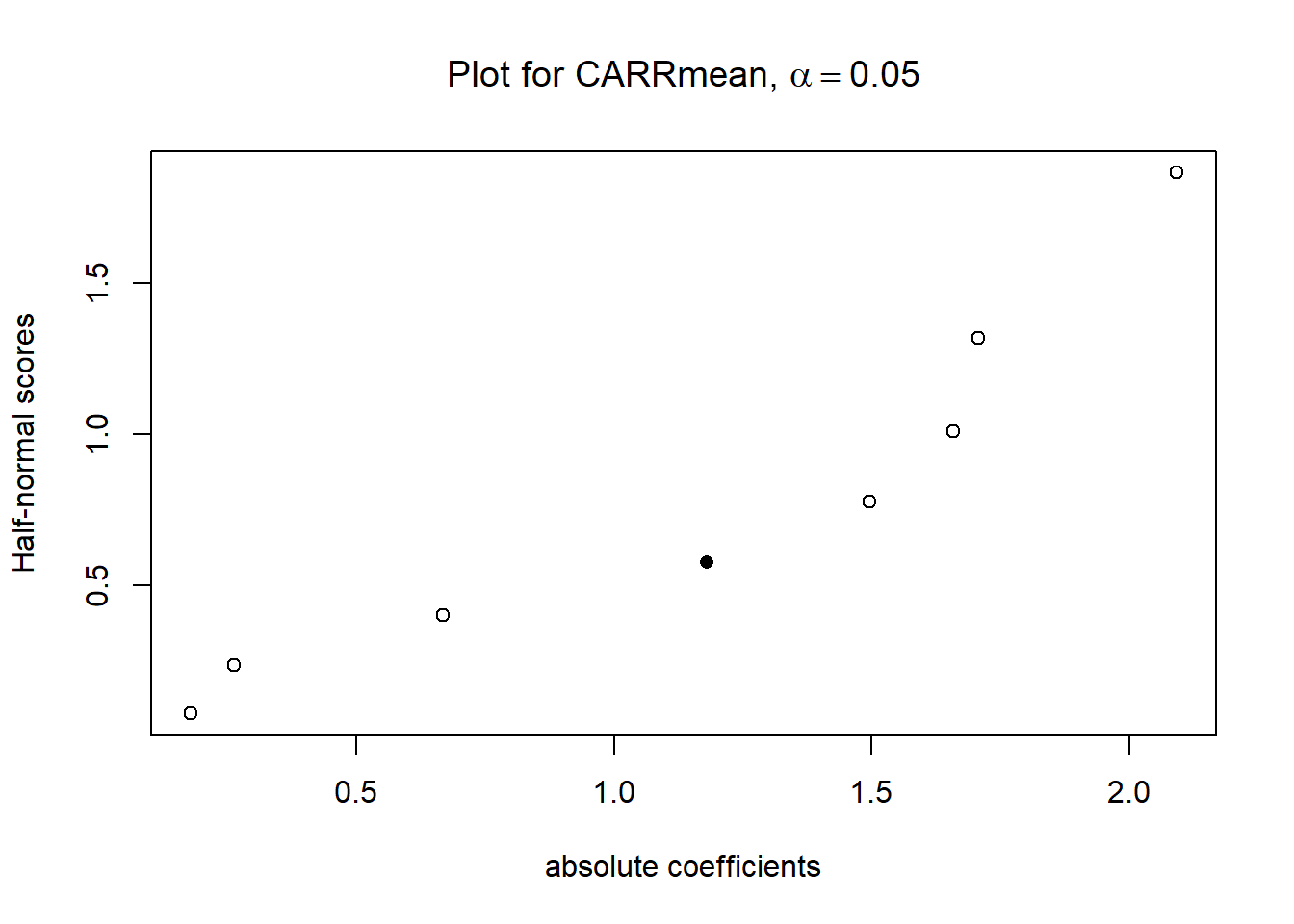
character(0)