12 Lewis 2001
El presente documento corresponde a un apartado del trabajo titulado “Guía para la implementación de diseños experimentales en la industria farmacéutica”, presentado por Nathalia Cortés Duque como requisito para optar al título de Magíster en Ciencias - Estadística de la Universidad Nacional de Colombia.
12.1 Ejemplo 1: Drill Data
Este es un diseño factorial \(2^4\) sin réplicas. El diseño es analizado usando tres modelos de regresión:
- Mínimos cuadrados ordinarios con los datos originales
- Mínimos cuadrados ordinarios después de transformar logarítmicamente los datos originales
- Modelo Lineal Generalizado asumiendo que la variable respuesta sigue una distribución Gamma y usando link de enlace logaritmo.
El diseño es el siguiente:
Código
creating full factorial with 16 runs ...12.1.1 Mínimos Cuadrados Ordinarios
Usando los datos originales, se ajusta el modelo lineal saturado (con todos los factores e interacciones).
Call:
lm.default(formula = y ~ A * B * C * D, data = dis_y)
Residuals:
ALL 16 residuals are 0: no residual degrees of freedom!
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 6.15500 NaN NaN NaN
A1 0.45625 NaN NaN NaN
B1 1.64875 NaN NaN NaN
C1 3.21625 NaN NaN NaN
D1 1.14250 NaN NaN NaN
A1:B1 0.07500 NaN NaN NaN
A1:C1 0.29750 NaN NaN NaN
B1:C1 0.75250 NaN NaN NaN
A1:D1 0.42125 NaN NaN NaN
B1:D1 0.22125 NaN NaN NaN
C1:D1 0.79875 NaN NaN NaN
A1:B1:C1 0.08375 NaN NaN NaN
A1:B1:D1 0.29500 NaN NaN NaN
A1:C1:D1 0.37750 NaN NaN NaN
B1:C1:D1 0.09000 NaN NaN NaN
A1:B1:C1:D1 0.26875 NaN NaN NaN
Residual standard error: NaN on 0 degrees of freedom
Multiple R-squared: 1, Adjusted R-squared: NaN
F-statistic: NaN on 15 and 0 DF, p-value: NADado que el modelo es saturado y no tiene réplicas, cada observación es predicha de forma perfecta por el modelo, no hay grados de libertad y por lo tanto no es posible calcular la varianza residual ni los p-valores asociados a los efectos de los factore y sus interacciones.
Aún así, es posible calcular los efectos estimados a partir del modelo de regresión ajustado. Si \(\beta\) es el coeficiente estimado del \(i\)-ésimo término del modelo de regresión, entonces su efecto estimado se calcula como \(2\times \beta\).
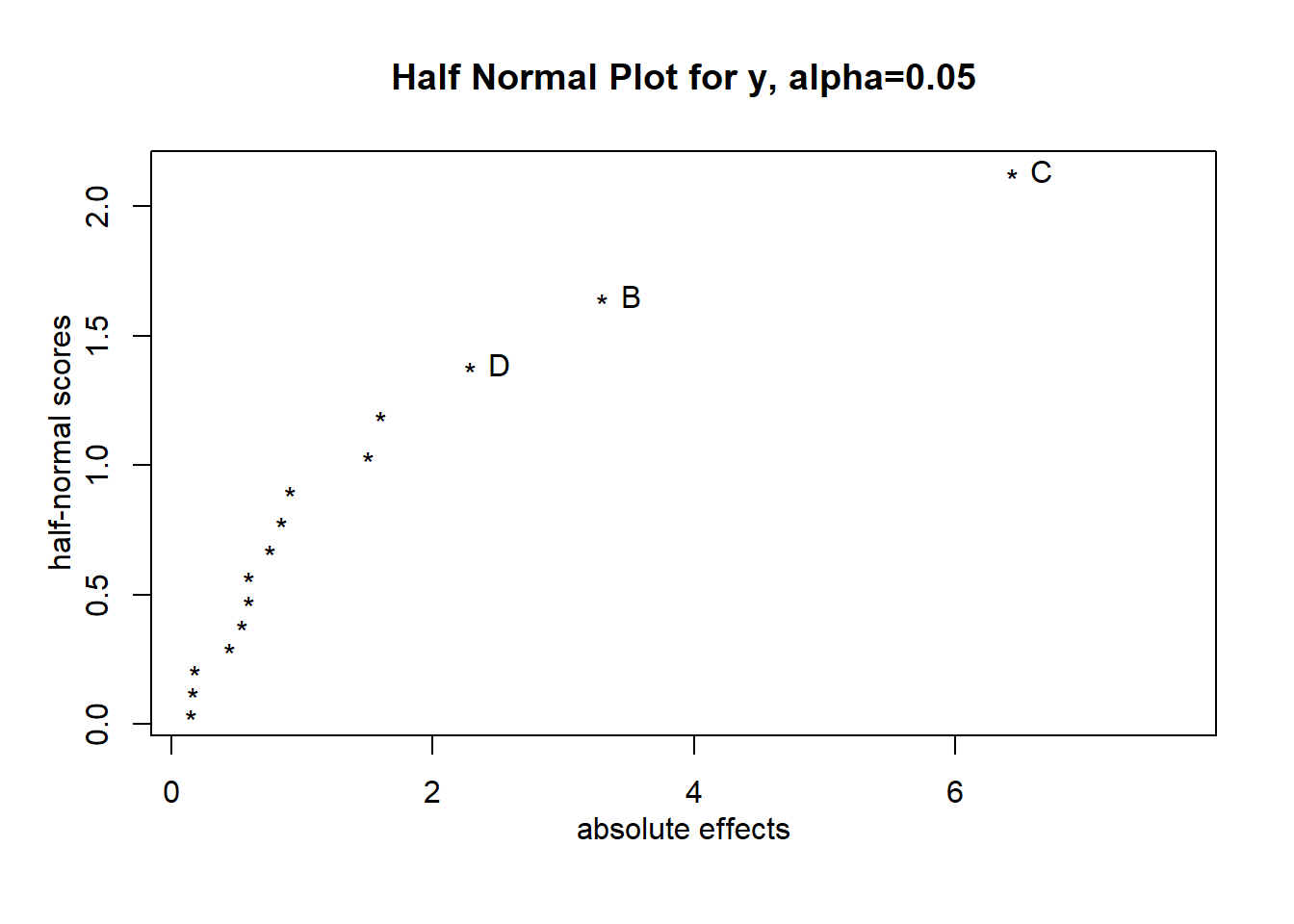
Dado que no es posible hacer un ANOVA, la significancia de los efectos puede ser aproximada por medio del Daniel Plot (half-normal plot), el cual identifica visualmente cuáles efectos son importantes (grandes).
- Se calculan todos los efectos principales e interacciones
- Se toma su valor absoluto
- Se ordenan del más pequeño al más grande
- Se grafican en un half-normal probability plot
- Eje x: estadísticas de orden esperadas de una distribución half-normal
- Eje Y: valores absolutos de los efectos
El gráfico es una guía sobre la magnitud de los efectos, pero no representa un test formal de significancia estadística. En este caso, los efectos de los factores \(x_2 (B), x_3 (C)\) y \(x_4 (D)\) son identificados como importantes según el Daniel Plot. Las interacciones \(x_2x_3\) y \(x_3x_4\) también están un poco desviadas de los efectos que están alineados, pero no fueron marcados por el Daniel Plot como importantes.
12.1.2 Transformación Logarítmica
Se transforma la variable respuesta con logaritmo y se crea el diseño.
Código
Call:
lm.default(formula = lny ~ A * B * C * D, data = dis_lny)
Residuals:
ALL 16 residuals are 0: no residual degrees of freedom!
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.597728 NaN NaN NaN
A1 0.064995 NaN NaN NaN
B1 0.290041 NaN NaN NaN
C1 0.577226 NaN NaN NaN
D1 0.163271 NaN NaN NaN
A1:B1 -0.017182 NaN NaN NaN
A1:C1 0.005176 NaN NaN NaN
B1:C1 -0.025102 NaN NaN NaN
A1:D1 0.033450 NaN NaN NaN
B1:D1 -0.007469 NaN NaN NaN
C1:D1 0.049070 NaN NaN NaN
A1:B1:C1 0.005187 NaN NaN NaN
A1:B1:D1 0.026108 NaN NaN NaN
A1:C1:D1 0.026613 NaN NaN NaN
B1:C1:D1 -0.017256 NaN NaN NaN
A1:B1:C1:D1 0.019308 NaN NaN NaN
Residual standard error: NaN on 0 degrees of freedom
Multiple R-squared: 1, Adjusted R-squared: NaN
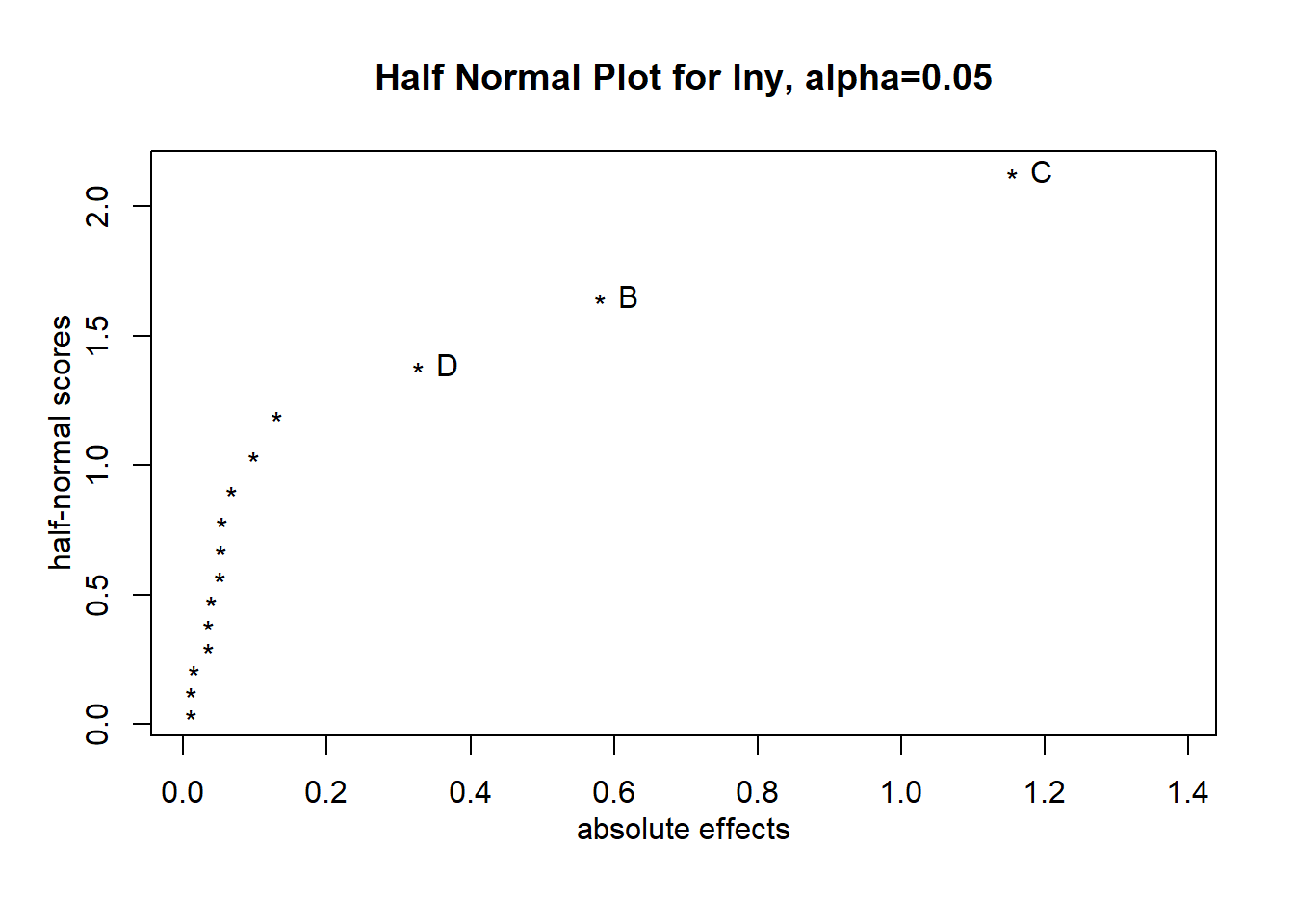
F-statistic: NaN on 15 and 0 DF, p-value: NAEl Daniel Plot sugiere que los factores importantes son \(x_2, x_3\) y \(x_4\).
Usando los factores sugeridos por el Daniel Plot, se ajusta el modelo y se hace el ANOVA, pues ahora sí hay grados de libertad para calcular el error residual.
Código
Call:
lm.default(formula = lny ~ B + C + D, data = dis_lny)
Residuals:
Min 1Q Median 3Q Max
-0.166184 -0.073140 0.000175 0.052479 0.195711
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.59773 0.03006 53.143 1.30e-15 ***
B1 0.29004 0.03006 9.647 5.27e-07 ***
C1 0.57723 0.03006 19.200 2.25e-10 ***
D1 0.16327 0.03006 5.431 0.000152 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.1203 on 12 degrees of freedom
Multiple R-squared: 0.9762, Adjusted R-squared: 0.9702
F-statistic: 163.7 on 3 and 12 DF, p-value: 5.339e-10Call:
aov.default(formula = mod3)
Terms:
B C D Residuals
Sum of Squares 1.345979 5.331036 0.426521 0.173544
Deg. of Freedom 1 1 1 12
Residual standard error: 0.1202582
Estimated effects are balancedLuego de ajustado el modelo de regresión, se obtuvo la siguiente ecuación de predicción:
\[\hat{y} = e^{1.5977 + 0.2900x_2 + 0.5772x_3 + 0.1633x_4}\]
A partir de esta ecuación, se obtienen las estimaciones de la media esperada de la variable respuesta para cada combinación de factores. Asimismo, se calcula el intervalo de confianza del \(95\%\) para cada estimación.
Código
# Matriz de diseño reducida y matriz especial
XRed <- model.matrix(mod3)
Mesp <- solve(t(XRed) %*% XRed)
# Parámetros para IC
sigma2 <- sigma(mod3)^2
t_crit <- qt(0.975, df = df.residual(mod3))
# Estimaciones y varianzas
estimates_log <- XRed %*% coef(mod3)
vars <- rowSums((XRed %*% Mesp) * XRed)
# IC en log y en escala original
se <- sqrt(sigma2 * vars)
ci_log <- cbind(
Lower = estimates_log - t_crit * se,
Estimate = estimates_log,
Upper = estimates_log + t_crit * se
)
ci <- exp(ci_log)
# Resultados finales
results <- round(cbind(ci_log, ci), 2)
colnames(results) <- c("Lower_log","Estimate_log","Upper_log",
"Lower","Estimate","Upper")
results_log <- as.data.frame(results)Código
Lower (log) | Estimate (log) | Upper (log) | Lower | Estimate | Upper |
|---|---|---|---|---|---|
0.44 | 0.57 | 0.70 | 1.55 | 1.76 | 2.01 |
0.44 | 0.57 | 0.70 | 1.55 | 1.76 | 2.01 |
1.02 | 1.15 | 1.28 | 2.76 | 3.15 | 3.59 |
1.02 | 1.15 | 1.28 | 2.76 | 3.15 | 3.59 |
1.59 | 1.72 | 1.85 | 4.91 | 5.59 | 6.38 |
1.59 | 1.72 | 1.85 | 4.91 | 5.59 | 6.38 |
2.17 | 2.30 | 2.43 | 8.76 | 9.99 | 11.39 |
2.17 | 2.30 | 2.43 | 8.76 | 9.99 | 11.39 |
0.76 | 0.89 | 1.02 | 2.14 | 2.44 | 2.79 |
0.76 | 0.89 | 1.02 | 2.14 | 2.44 | 2.79 |
1.34 | 1.47 | 1.60 | 3.83 | 4.37 | 4.98 |
1.34 | 1.47 | 1.60 | 3.83 | 4.37 | 4.98 |
1.92 | 2.05 | 2.18 | 6.80 | 7.75 | 8.84 |
1.92 | 2.05 | 2.18 | 6.80 | 7.75 | 8.84 |
2.50 | 2.63 | 2.76 | 12.15 | 13.85 | 15.79 |
2.50 | 2.63 | 2.76 | 12.15 | 13.85 | 15.79 |
12.1.3 Modelo Lineal Generalizado
Se ajusta un Modelo Lineal Generalizado (MLG) asumiendo que la variable respuesta tiene una distribución Gamma, y se usa la función de enlace Logaritmo.
Warning in dgamma(y, 1/disp, scale = mu * disp, log = TRUE): Se han producido
NaNs
Call:
glm(formula = y ~ A * B * C * D, family = Gamma(link = "log"),
data = dis_y)
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.597728 NaN NaN NaN
A1 0.064995 NaN NaN NaN
B1 0.290041 NaN NaN NaN
C1 0.577226 NaN NaN NaN
D1 0.163271 NaN NaN NaN
A1:B1 -0.017182 NaN NaN NaN
A1:C1 0.005176 NaN NaN NaN
B1:C1 -0.025102 NaN NaN NaN
A1:D1 0.033450 NaN NaN NaN
B1:D1 -0.007469 NaN NaN NaN
C1:D1 0.049070 NaN NaN NaN
A1:B1:C1 0.005187 NaN NaN NaN
A1:B1:D1 0.026108 NaN NaN NaN
A1:C1:D1 0.026613 NaN NaN NaN
B1:C1:D1 -0.017256 NaN NaN NaN
A1:B1:C1:D1 0.019308 NaN NaN NaN
(Dispersion parameter for Gamma family taken to be NaN)
Null deviance: 7.0252e+00 on 15 degrees of freedom
Residual deviance: -5.4349e-16 on 0 degrees of freedom
AIC: NaN
Number of Fisher Scoring iterations: 1Analysis of Deviance Table
Model: Gamma, link: log
Response: y
Terms added sequentially (first to last)
Df Deviance Resid. Df Resid. Dev
NULL 15 7.0252
A 1 0.0882 14 6.9370
B 1 1.1967 13 5.7403
C 1 5.2109 12 0.5293
D 1 0.4241 11 0.1053
A:B 1 0.0047 10 0.1006
A:C 1 0.0006 9 0.1000
B:C 1 0.0093 8 0.0907
A:D 1 0.0177 7 0.0729
B:D 1 0.0009 6 0.0721
C:D 1 0.0387 5 0.0334
A:B:C 1 0.0004 4 0.0329
A:B:D 1 0.0109 3 0.0221
A:C:D 1 0.0113 2 0.0107
B:C:D 1 0.0048 1 0.0060
A:B:C:D 1 0.0060 0 0.0000Código
(Intercept) C1 B1 D1 A1 C1:D1
1.597728288 0.577225902 0.290040837 0.163271482 0.064995204 0.049070479
A1:D1 A1:C1:D1 A1:B1:D1 A1:B1:C1:D1 A1:B1:C1 A1:C1
0.033450321 0.026613408 0.026108317 0.019308000 0.005186800 0.005175516
B1:D1 A1:B1 B1:C1:D1 B1:C1
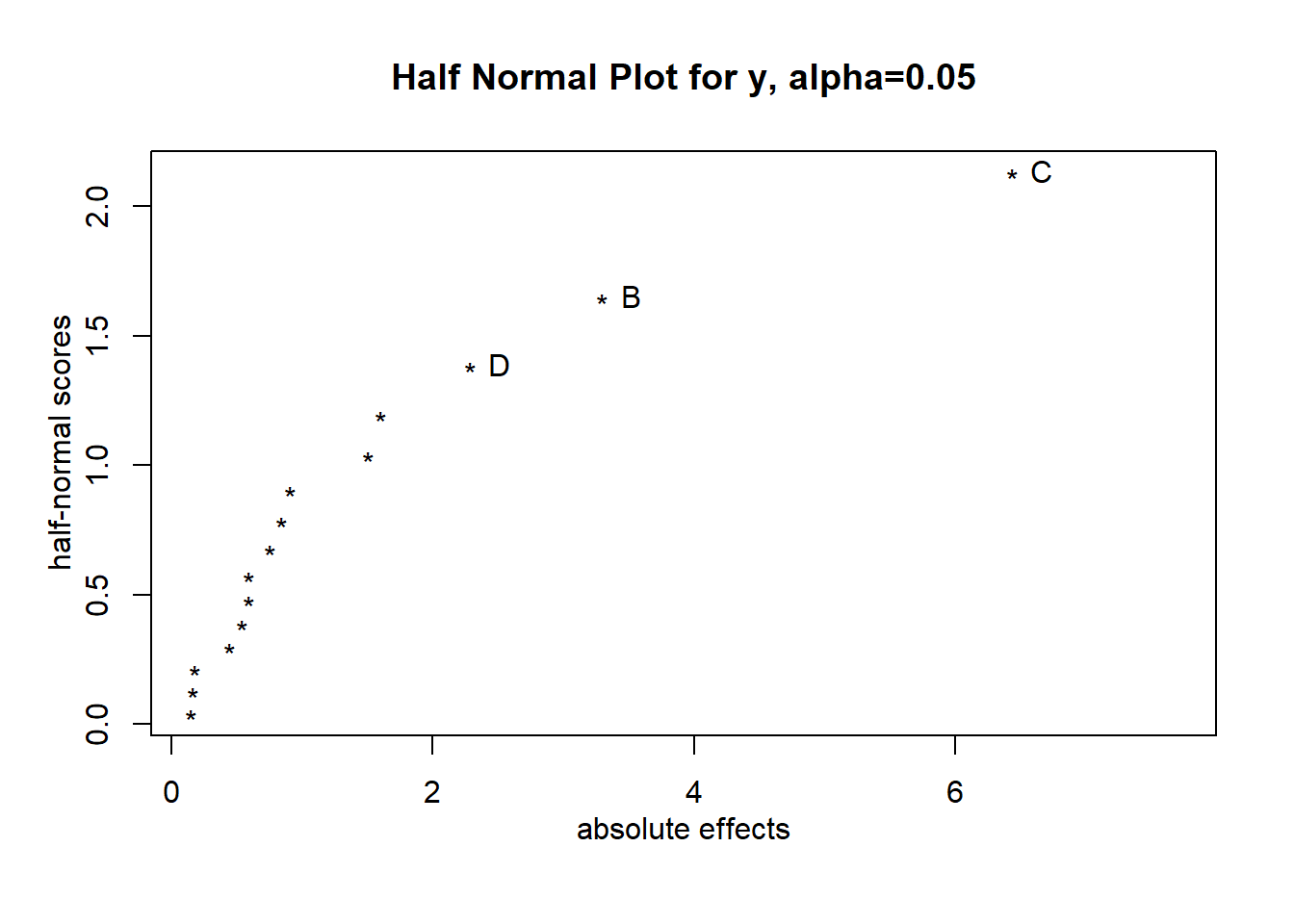
-0.007469293 -0.017181633 -0.017256294 -0.025102228 En este caso, el Daniel Plot también sugiere que los factores más importantes son \(x1, x2\) y \(x3\).
Se ajusta entonces el modelo MLG usando los factores \(x_1, x_2\) y \(x_3\).
Call:
glm(formula = y ~ B + C + D, family = Gamma(link = "log"), data = dis_y)
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.60317 0.03021 53.074 1.32e-15 ***
B1 0.28952 0.03021 9.585 5.65e-07 ***
C1 0.57894 0.03021 19.166 2.29e-10 ***
D1 0.16560 0.03021 5.482 0.00014 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for Gamma family taken to be 0.01459884)
Null deviance: 7.02517 on 15 degrees of freedom
Residual deviance: 0.17404 on 12 degrees of freedom
AIC: 34.225
Number of Fisher Scoring iterations: 4Analysis of Deviance Table
Model: Gamma, link: log
Response: y
Terms added sequentially (first to last)
Df Deviance Resid. Df Resid. Dev F Pr(>F)
NULL 15 7.0252
B 1 1.1914 14 5.8338 81.606 1.061e-06 ***
C 1 5.2241 13 0.6097 357.844 2.669e-10 ***
D 1 0.4357 12 0.1740 29.842 0.0001447 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1La ecuación de predicción obtenida en este caso es como sigue:
\[\hat{y} = e^{1.6032 + 0.2895x_2 + 0.5789x_3 + 0.1656x_4}\]
Los intervalos de confianza del \(95\%\) para la media condicional de las respuestas se calcularon mediante la fórmula de Wald para la distribución Gamma con enlace logarítmico, tal como se describe en Lewis (2001).
\[ e^{x_i' \hat{\beta}} \;\pm\; z_{\alpha/2} \, e^{x_i' \hat{\beta}} \, \nu \sqrt{\, x_i' (X'X)^{-1} x_i \,} \]
Código
X <- model.matrix(modg2)
eta_hat <- as.numeric(X %*% coef(modg2))
mu_hat <- exp(eta_hat) # keep full precision
M <- vcov(modg2)
se_eta <- sqrt(rowSums((X %*% M) * X))
z <- qnorm(0.975)
lower <- mu_hat - z * mu_hat * se_eta
upper <- mu_hat + z * mu_hat * se_eta
results_mlg <- data.frame(Lower = as.numeric(lower),
Estimate = as.numeric(mu_hat),
Upper = as.numeric(upper)) %>%
round(2)Código
Lower | Estimate | Upper |
|---|---|---|
1.56 | 1.77 | 1.98 |
1.56 | 1.77 | 1.98 |
2.78 | 3.15 | 3.53 |
2.78 | 3.15 | 3.53 |
4.96 | 5.62 | 6.29 |
4.96 | 5.62 | 6.29 |
8.85 | 10.03 | 11.22 |
8.85 | 10.03 | 11.22 |
2.17 | 2.46 | 2.75 |
2.17 | 2.46 | 2.75 |
3.87 | 4.39 | 4.91 |
3.87 | 4.39 | 4.91 |
6.90 | 7.83 | 8.76 |
6.90 | 7.83 | 8.76 |
12.32 | 13.97 | 15.63 |
12.32 | 13.97 | 15.63 |
12.1.4 Resultados
A continuación se muestran los efectos estimados del modelo saturado usando los tres modelos: MCO con los datos originales, MCO con los datos transformados con logaritmo y el modelo MLG.
Código
# Nombres de los efectos
efectos_nombres <- c(
"Intercepto",
"x1", "x2", "x3", "x4",
"x1:x2", "x1:x3", "x2:x3", "x1:x4", "x2:x4", "x3:x4",
"x1:x2:x3", "x1:x2:x4", "x1:x3:x4", "x2:x3:x4",
"x1:x2:x3:x4"
)
tabla_efectos <- data.frame(
Efecto = efectos_nombres,
Efectos1 = round(efectos1, 3),
Efectos2 = round(efectos2, 3),
Efectos3 = round(efectos3, 3)
)Código
Efecto | Datos originales | Transformación log | MLG |
|---|---|---|---|
Intercepto | 6.155 | 1.598 | 1.598 |
x1 | 0.912 | 0.130 | 0.130 |
x2 | 3.298 | 0.580 | 0.580 |
x3 | 6.433 | 1.154 | 1.154 |
x4 | 2.285 | 0.327 | 0.327 |
x1:x2 | 0.150 | -0.034 | -0.034 |
x1:x3 | 0.595 | 0.010 | 0.010 |
x2:x3 | 1.505 | -0.050 | -0.050 |
x1:x4 | 0.843 | 0.067 | 0.067 |
x2:x4 | 0.442 | -0.015 | -0.015 |
x3:x4 | 1.598 | 0.098 | 0.098 |
x1:x2:x3 | 0.167 | 0.010 | 0.010 |
x1:x2:x4 | 0.590 | 0.052 | 0.052 |
x1:x3:x4 | 0.755 | 0.053 | 0.053 |
x2:x3:x4 | 0.180 | -0.035 | -0.035 |
x1:x2:x3:x4 | 0.537 | 0.039 | 0.039 |
Los efectos estimados obtenidos con el modelo MCO con los datos transformados y el modelo MLG son los mismos.
Para evaluar la precisión de las estimaciones del modelo, puede analizarse la amplitud de los intervalos de confianza. Una menor amplitud indica mayor precisión en las estimaciones, mientras que intervalos más amplios reflejan mayor incertidumbre.
Código
Amplitud_Log | Amplitud_MLG |
|---|---|
0.46 | 0.42 |
0.46 | 0.42 |
0.83 | 0.75 |
0.83 | 0.75 |
1.47 | 1.33 |
1.47 | 1.33 |
2.63 | 2.37 |
2.63 | 2.37 |
0.65 | 0.58 |
0.65 | 0.58 |
1.15 | 1.04 |
1.15 | 1.04 |
2.04 | 1.86 |
2.04 | 1.86 |
3.64 | 3.31 |
3.64 | 3.31 |
Se observa que la amplitud de los intervalos de confianza de las estimaciones obtenidas con el modelo MLG es menor que la del modelo MCO con transformación logarítmica, lo que refleja una mayor precisión en la estimación de la media.